«`html
Модели машинного обучения для прогнозирования эффективности редактирования Prime
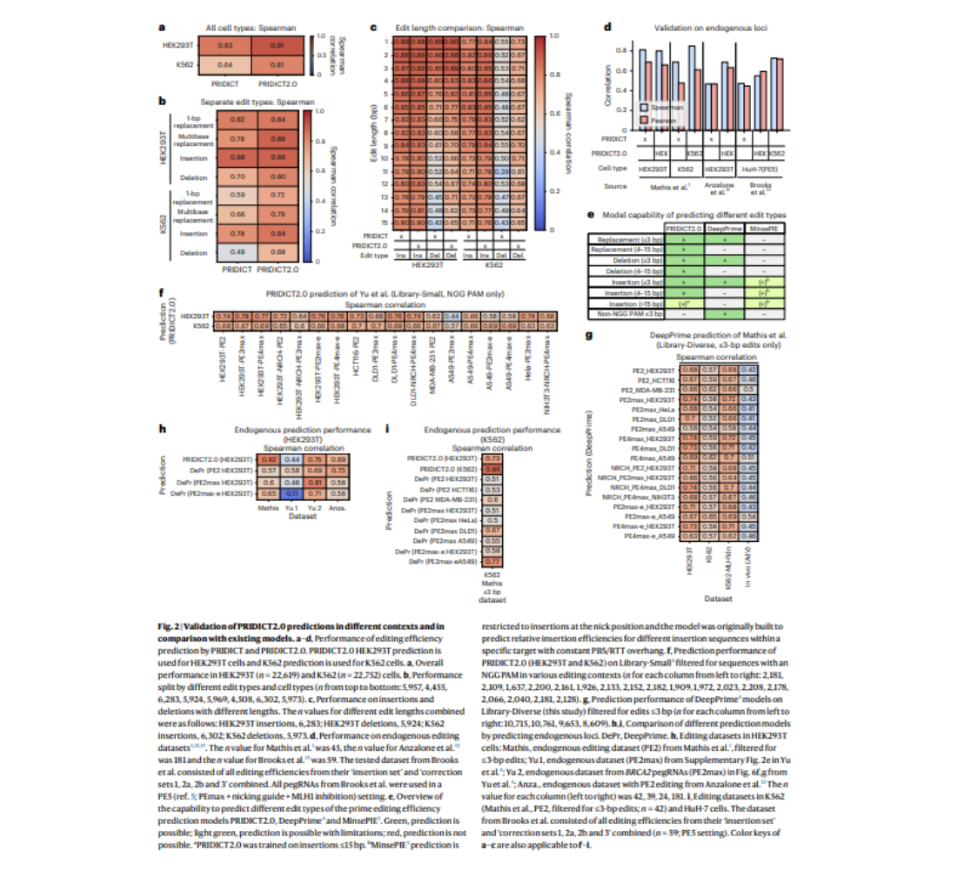
Успех редактирования Prime в значительной степени зависит от конструкции прайм-редактирующего РНК-гида (pegRNA) и местоположения целевого локуса. Для решения этой проблемы исследователи разработали две взаимодополняющие модели машинного обучения — PRIDICT2.0 и ePRIDICT — для прогнозирования эффективности редактирования Prime в различных типах редактирования и хроматиновых контекстах. PRIDICT2.0, усовершенствованная версия предыдущей модели PRIDICT1, оценивает производительность pegRNA для редактирования до 15 пар оснований (bp) в клеточных линиях с нарушением и без нарушения ремонта несоответствий (MMR). В то же время ePRIDICT количественно оценивает, как местные хроматиновые окружения влияют на скорость редактирования Prime. Используя разнообразную библиотеку pegRNA в клетках HEK293T (с нарушением MMR) и K562 (без нарушения MMR), исследование показало, что PRIDICT2.0 значительно превосходит своего предшественника, особенно в случае многозаместительных замен и удалений. Устойчивость модели была подтверждена через обширную валидацию, показав сильные корреляции между экспериментальными повторами и улучшенную производительность по сравнению с предыдущими моделями.
Понимание хроматинового контекста и эффективности редактирования
Одним из ключевых достижений этого исследования является включение хроматинового контекста как фактора, влияющего на эффективность редактирования Prime. ePRIDICT была разработана для прогнозирования результатов редактирования, учитывая локус-специфические хроматиновые особенности, добавляя новый уровень точности к прогнозам редактирования. Анализ объяснений Шэпли (SHAP) показал, что такие характеристики, как длина редактирования, наличие последовательностей полиТ и длина RTT оверхэнга, были высоко значимы в клетках HEK293T, в то время как позиция, температура плавления и содержание G+C играли решающую роль в клетках K562. Исследование также показало, что паттерны редактирования в клетках с нарушением MMR напоминали те, что в клетках K562 с подавленными путями MMR, что еще раз подчеркивает важность учета хроматинового контекста для точных прогнозов. На основе этих результатов модели предлагают ценные инструменты для улучшения конструкции pegRNA и максимизации эффективности редактирования Prime в различных биологических контекстах.
Роль хроматина в эффективности редактирования генома
Для изучения влияния хроматина на редактирование генома клетки были обработаны ABE8e, BE4max и Cas9, что показало сильные корреляции в эффективности редактирования, особенно между ABE8e и BE4max. Активные хроматиновые особенности, такие как ATAC-Seq и H3K4me3, положительно коррелировали с эффективностью редактирования, в то время как подавляющие метки (H3K9me3, H3K27me3) были связаны с более низкой эффективностью. Анализ UMAP выделил хроматиновый градиент, влияющий на редактирование. Основанная на XGBoost модель ‘ePRIDICT’, обученная на хроматиновых данных, эффективно прогнозировала результаты редактирования. Комбинирование ее с PRIDICT2.0 повысило точность, особенно в регионах с более низкой доступностью хроматина, подтверждая решающую роль хроматина в результатах редактирования.
Клонирование и конструкция pegRNA
Библиотека плазмид TRIP, использованная в исследованиях хроматинового контекста, была создана в соответствии с определенным протоколом. Для валидации pegRNA на эндогенных мишенях было выбрано 20 геномных сайтов из предыдущего скрининга — 10 сайтов с высокой и 10 с низкой эффективностью редактирования. PegRNA были разработаны для достижения различных генетических модификаций: замены 1 пары оснований, вставки 4 пар оснований и удаления 4 пар оснований. PegRNA были выбраны на основе их прогнозируемой эффективности редактирования и конкретного наличия нуклеотидов в их целевых окнах. Кроме того, было разработано и склонировано еще 90 pegRNA, нацеленных на интронные и межгенные регионы, с использованием определенного вектора. sgRNA были введены в плазмиду через реакцию клонирования в одну стадию, затем трансформированы в компетентные бактериальные клетки, произведена экстракция и верификация плазмид.
Производство и скрининг вирусных векторов
Лентивирусные и псевдотипированные векторы AAV9 были произведены путем трансфекции клеток HEK293T необходимыми плазмидами и очистки векторов через серию осаждения и центрифугирования. Также был произведен отдельный вирусный вектор, содержащий компонент редактирования Prime. Библиотека pegRNA, разработанная для включения патогенных вариантов и мутаций в некодирующих областях, была заказана у коммерческого поставщика. Различные клеточные линии, включая HEK293T, HepG2 и K562, поддерживались в специфических условиях и подвергались трансфекции или электропорации для редактирования. Скрининг включал трансдукцию клеток лентивирусом и выбор отредактированных клеток с использованием антибиотиков. Для in vivo исследований векторы инъецировали мышам, которых затем усыпляли для изоляции гепатоцитов. Геномная ДНК из этих экспериментов была изолирована и проанализирована с использованием техник секвенирования высокой пропускной способности.
Анализ библиотеки и эффективности редактирования
Последовательности чтения были обрезаны и отфильтрованы для обеспечения точности, удаляя ~34% чтений в клетках HEK293T и K562 и ~60% в гепатоцитах мыши. Эффективность редактирования была рассчитана путем сравнения последовательностей чтения с диким типом и отредактированными последовательностями, корректируя фоновые частоты. PegRNA были валидированы с использованием конкретных критериев и усреднены по повторам, что привело к нескольким наборам данных. Для библиотеки TRIP, тагментация была сопровождена ПЦР-усилением и секвенированием. Эффективность редактирования была проанализирована с помощью специальных скриптов и перекрестно сопоставлена с хроматиновыми данными из ENCODE. Модели машинного обучения, включая PRIDICT2.0, были обучены и валидированы с использованием различных наборов данных, а их производительность оценивалась путем перекрестной валидации и анализа важности признаков.
Check out the Paper. All credit for this research goes to the researchers of this project. Also, don’t forget to follow us on Twitter and join our Telegram Channel and LinkedIn Group. If you like our work, you will love our newsletter..
Don’t Forget to join our 50k+ ML SubReddit
FREE AI WEBINAR: ‘SAM 2 for Video: How to Fine-tune On Your Data’ (Wed, Sep 25, 4:00 AM – 4:45 AM EST)
The post Advancements in Machine Learning Models and Chromatin Context for Optimizing Prime Editing Efficiency appeared first on MarkTechPost.
«`




















